在Python中对相关矩阵进行排序
相关矩阵在同一时间调查多个变量之间的依赖关系。它显示了对称的表格数据,其中每一行和每一列代表一个变量,相应的值是相关系数,表示这两个变量之间的关系强度。有许多类型的相关系数(皮尔逊系数、肯德尔系数、斯皮尔曼系数等),它们是由不同的相关分析方法计算出来的。相关系数值接近1的变量显示出强烈的正相关,接近-1的变量显示出强烈的负相关,接近0的变量显示出弱相关或无相关。
在数据分析中,相关矩阵对于总结和发现大量数据中的关系非常有用。它也是机器学习中探索性数据分析和特征选择的一个常用指标。
解释一个相关矩阵在大数据中可能会变得很困难。有时,对相关值进行排序有助于轻松看到各种变量对的依赖程度。在这篇文章中,我们将看到如何用Python对相关矩阵进行排序。
步骤
- Import module
- Load data
- 使用上述数据创建一个相关矩阵
- 对数据进行分类。
- 显示排序后的数据
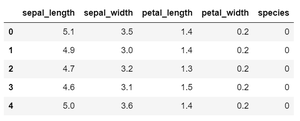
我们将使用Python的Seaborn包中的鸢尾花数据集。该数据集包含一种鸢尾花的3个类,每个类有50个属性实例。注意,相关矩阵忽略了数据中任何非数字列。因此,首先使用标签编码将你想包含在你的相关矩阵中的任何非数字数据改为数字数据。
现在,为了对相关矩阵进行排序,首先我们必须将矩阵转换为一维序列。unstack()函数被用来这样做。该系列将有多个索引。
为了进行排序,使用了sort_values()函数。sort_values()函数按照所传递的Column的升序或降序对一个数据框进行排序。
语法:DataFrame.sort_values(by, axis=0, ascending=True, inplace=False, kind='quicksort', na_position='last')
参数:
- by:单一的/列表的列名来排序数据框架。
- axis:0或’index’代表行,1或’column’代表列。
- ascending:如果为真,则以升序排列数据框架的布尔值。
- inplace:Boolean值。如果为真,则在传递的数据框中进行更改。
- kind:可以有三个输入(‘quicksort’、’mergesort’或’heapsort’)的用于排序数据框的算法的_字符串。
- na_position:接受两个字符串输入’last’或’first’来设置Null值的位置。默认为 “最后”。
返回类型。返回一个排序的数据框,其尺寸与函数调用者的数据框相同。_
使用中的数据框架:
示例 1:
# Import required libraries
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.preprocessing import LabelEncoder
# Load dataset
df = sns.load_dataset('iris')
# Convert categorical values to numeric
label_encoder = LabelEncoder()
df['species'] = label_encoder.fit_transform(df['species'])
# Create correlation matrix
corr_mat = df.corr(method='pearson')
# Convert correlation matrix to 1-D Series and sort
sorted_mat = corr_mat.unstack().sort_values()
print(sorted_mat)
输出:
例子2:对相关矩阵进行排序,没有重复的。
为了去除重复的和自相关的值,在将相关矩阵转换为一维数列之前获得矩阵的上三角或下三角值。为此目的,使用了triu()函数,它返回一个具有相关矩阵形状的上三角矩阵(主对角线以上的元素的值为1,其他为0)。astype()方法将矩阵值转换为布尔值。这时,函数可以根据另一个条件数组来选择数组,所以我们得到的结果是一个具有相关矩阵上三角值的矩阵,其余的值为空。
然后将相关矩阵转换为一维数组进行排序,就像上面的例子那样。下面给出了实现方法。
# Import required libraries
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.preprocessing import LabelEncoder
# Load dataset
df = sns.load_dataset('iris')
# Convert categorical values to numeric
label_encoder = LabelEncoder()
df['species'] = label_encoder.fit_transform(df['species'])
# Create correlation matrix
corr_mat = df.corr(method='pearson')
# Retain upper triangular values of correlation matrix and
# make Lower triangular values Null
upper_corr_mat = corr_mat.where(
np.triu(np.ones(corr_mat.shape), k=1).astype(np.bool))
# Convert to 1-D series and drop Null values
unique_corr_pairs = upper_corr_mat.unstack().dropna()
# Sort correlation pairs
sorted_mat = unique_corr_pairs.sort_values()
print(sorted_mat)
输出:
 极客教程
极客教程