用Python将协方差矩阵转换为相关矩阵
在这篇文章中,我们将讨论协方差和相关性之间的关系,并使用python编程我们自己的函数来计算协方差和相关性。
协方差
它告诉我们两个量是如何相互关联的,比如我们想计算x和y之间的协方差,那么结果可以是其中之一。
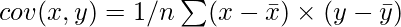
其中,
分别是x和y的平均值。
解释输出。
要么x和y之间的协方差是 。
协方差(x,y)>0:这意味着它们是正相关的。
协方差(x,y)< 0:这意味着x和y是负相关的。
如果Covariance(x,y) = 0 : 那么x和y是相互独立的。
协方差矩阵:
协方差提供了一个衡量两个变量或更多变量集之间相关性的强度,为了计算协方差矩阵,使用了numpy的cov()方法。
语法:
ny.cov(m, y=None, rowvar=True, bias=False, ddof=None, fweights=None, aweights=None **) **
m : [array_like] 一个一维或二维的变量。变量是列
y : [array_like] 它与m的形式相同。
rowvar : [bool, optional] 如果rowvar为True(默认),那么每一行代表一个变量,列中有观测值。否则,这种关系将被转置。
bias : 默认的规范化是假的。如果bias为True,它将对数据点进行归一化处理。
ddof : 如果不是无,那么bias所暗示的默认值将被覆盖。请注意,ddof=1将返回无偏估计,即使同时指定了fweights和aweights。
fweights : fweight是整数频率权重的一维数组。
aweights : aweight是观察向量权重的一维数组。
返回:它返回ndarray协方差矩阵
相关性
它显示了成对的变量之间是否有关系以及有多大的关系。相关性的数值在-1到+1之间,其中接近+1的数值代表强正相关,接近-1的数值代表强负相关。它给出了变量之间关系的方向和强度。
相关矩阵:
它基本上是一个协方差矩阵。也被称为自动协方差矩阵,分散矩阵,方差矩阵,或方差协方差矩阵。它是一个矩阵,其中i-j位置定义了给定数据集的i th _和_j th _参数之间的相关性。它使用_numpy的corrcoeff()方法计算。
语法:
numpy.corrcoef(x, y=None, rowvar=True, bias=<no value>, ddof=<no value>)
x :一个包含多个变量和观测值的一维或二维数组。x的每一行代表一个变量,每一列代表所有这些变量的一个观测值。也见下面的rowvar。
y , 可选。一组额外的变量和观测值。y的形状与x相同。
rowvar : 如果rowvar为True(默认),那么每一行代表一个变量,列中有观测值。否则,这种关系是换位的:每一列代表一个变量,而行则包含观测值。
返回: ndarray
那么,为什么我们需要关联性?
1.协方差告诉我们两个随机变量是+V还是-V的关系,但并没有告诉我们是多少。
2.协方差是对算术变化的变体,例如:如果我们把x乘以10或除以10,那么结果就会发生变化,这对相关来说是不对的,因为相关的结果在这种操作中保持不变。
3.协方差的输出是很难比较的,因为其数值范围可以从-无穷大到+无穷大。而相关度的输出值从0到1不等。
相关和协方差之间的关系
相关性只是归一化的协方差,请参考以下公式。
其中,
分别为x和y的标准差。
将协方差矩阵转换为相关矩阵的Python程序
为了解决这个问题,我们选择了虹膜数据,因为要计算协方差,我们需要数据,如果我们使用一个真实的例子数据集会更好。
加载和显示数据集
import numpy as np
import pandas as pd
# loading in the iris dataset for demo purposes
dataset = pd.read_csv("iris.csv")
dataset.head()
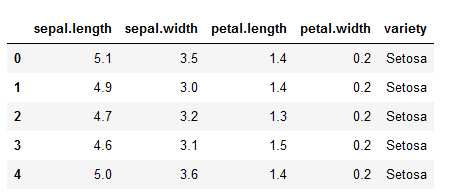
在这个例子中,我们将不使用目标列
data = dataset.iloc[:, :-1].values
实现协方差矩阵的程序:
# calculates the covariance between x and y
def calcCov(x, y):
mean_x, mean_y = x.mean(), y.mean()
n = len(x)
return sum((x - mean_x) * (y - mean_y)) / n
# calculates the Covariance matrix
def covMat(data):
# get the rows and cols
rows, cols = data.shape
# the covariance matroix has a shape of n_features x n_features
# n_featurs = cols - 1 (not including the target column)
cov_mat = np.zeros((cols, cols))
for i in range(cols):
for j in range(cols):
# store the value in the matrix
cov_mat[i][j] = calcCov(data[:, i], data[:, j])
return cov_mat
covMat(data)
输出 :
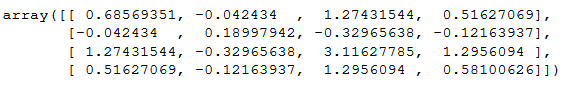
Numpy cov()输出:
np.cov(data,rowvar=False)
注意:rowVars需要设置为false,否则它将把行作为特征,把列作为观察值。
输出 :

计算相关度:
在这个函数中,我们将把协方差矩阵转换为相关关系。
# Now calculating Correlation using our Covariance function (covMat())
def corrMat(data):
rows, cols = data.shape
corr_mat = np.zeros((cols, cols))
for i in range(cols):
for j in range(cols):
x, y = data[:, i], data[:, j]
# not here that we are just normalizing the covariance matrix
corr_mat[i][j] = calcCov(x, y) / (x.std() * y.std())
return corr_mat
corrMat(data)
输出:

numpy中的corrcoef()也可以用来计算相关关系。
np.corrcoef(data,rowvar=False)
输出:

 极客教程
极客教程

