R语言 使用ggplot2的误差条
误差条是显示平均分数的条形。误差条像胡须一样从条形图中伸出来 。误差条显示了测量的精确程度。它显示了我们得到的数值有多少预期的变化。误差条可以在水平和垂直方向上镀上一层。水平误差条图显示了小组差异的误差条以及小组的条形图。
误差条以3种方式之一显示平均值的精确度。
- 置信区间
- 平均数的标准误差
- 标准偏差
geom_errorbar()
各种表示垂直区间的方式由x、ymin和ymax定义。每种情况都会画出一个图形对象。这里geom画的是误差条,它可以由下限和上限的值来定义。 记住,你必须自己提供y_min和y_max的值,因为geom的误差条不会自动计算置信度。
语法:
geom_errorbar(mapping = NULL,data = NULL,stat = "identity",position = "identity",na.rm = FALSE, orientation = NA,show.legend = NA, inherit.ais = TRUE)
例子:
library("ggplot2")
df <- ToothGrowth
# Transform dose and len column into factor
dfdose <- as.factor(dfdose)
dflen <- as.factor(dflen)
p<-ggplot(df,aes(dose,len))
p +
geom_line(aes(group = len/2))+geom_errorbar(
aes(ymin =len , ymax = dose),width=0.3)
输出:
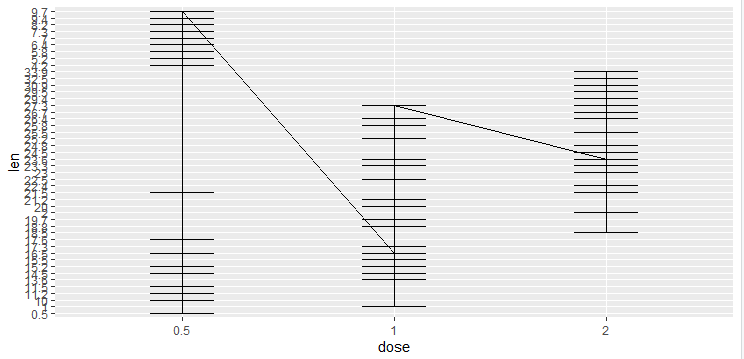
geom_linerange()
在这里,geom画出了误差条,它可以由下限和上限的值来定义。linerange函数与误差条有点类似。在直线范围函数中,你必须自己提供y_min和y_max的值,因为linerange geom并不自动计算置信度。在 geom_linerange 中,有一些参数是默认存在的(大小、行距、颜色、宽度)。
语法:
geom_linerange(mapping = NULL,data = NULL,stat = "identity",position = "identity",na.rm = FALSE orientation = NA,show.legend = NA,inherit.aes = TRUE)
例子:
# import libraries
library("ggplot2")
# import data
df <- ToothGrowth
# Transform dose and len column into factor
dfdose <- as.factor(dfdose)
dflen <- as.factor(dflen)
p<-ggplot(df,aes(dose,len))
p +
geom_line()+
stat_summary(func.y= mean, geom= "bar" ,fill="white",color="black") +
geom_linerange(aes(ymin =len , ymax = dose)
)
输出:
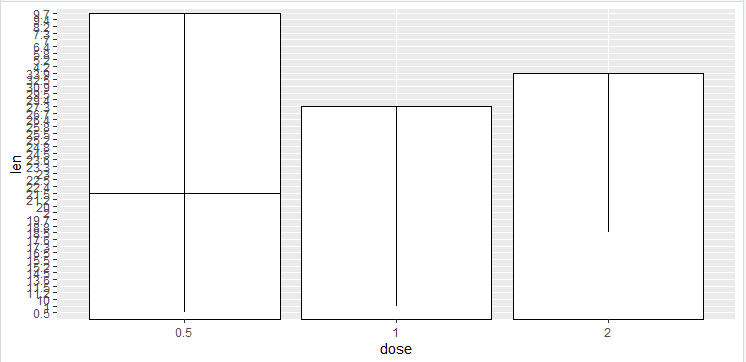
geom_pointrange()
在这里,geom画出了误差条,它可以由下限和上限的值来定义。点范围函数与误差条和线范围很相似。在点范围函数中,你必须自己提供y_min和y_max的值,因为geom的pointrange不会自动计算置信度。在geom_pointrange中,有一些参数是默认存在的(大小、线条范围、颜色、填充、宽度)。pointrange 函数对于绘制置信区间非常有用。
语法:
geom_linerange(mapping = NULL,data = NULL,stat = "identity",position = "identity",fatten = 4,na.rm = FALSE orientation = NA,show.legend = NA, inherit.ais = TRUE)
例子:
# import libraries
library("ggplot2")
# import data
df <- ToothGrowth
# Transform dose and len column into factor
dfdose <- as.factor(dfdose)
dflen <- as.factor(dflen)
p<-ggplot(df,aes(dose,len))
p +
geom_line()+
stat_summary(func.y= mean, geom= "bar" ,fill="white",color="black") +
geom_pointrange(aes(ymin =len , ymax = dose)
)
输出:
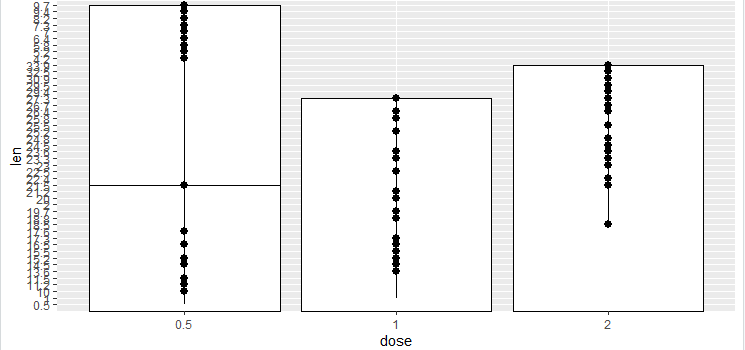
geom_crossbar()
在这里,geom画出了误差条,它可以由下限和上限的值来定义。crossbar函数与误差条和直线范围相当类似。交叉条是中间有水平线的空心条。
语法
geom_crossbar(mapping = NULL,data = NULL,stat = "identity",position = "identity",fatten = 2.5,na.rm = FALSE,orientation = NA,show.legend = NA, inherit.ais = TRUE)
例子:
# import libraries
library("ggplot2")
# import data
df <- ToothGrowth
# Transform dose and len column into factor
dfdose <- as.factor(dfdose)
dflen <- as.factor(dflen)
p<-ggplot(df,aes(dose,len))
p +geom_crossbar(aes(ymin = len , ymax = dose),width = 0.2)
输出:

geom_errorbarh()
在这里,geom画出了误差条,它可以由下限和上限的值来定义。geombar函数与误差条有点类似,但这里我们改变的是位置和轴。在errorbar范围函数中,你必须自己提供x_min和x_max的值,因为geom的误差条不会自动计算置信度。在geom_errorbar中,有一些参数是默认存在的(大小、线条范围、颜色、宽度)。
语法
geom_errorbarh(mapping = NULL,data = NULL,stat = "identity",position = "identity",na.rm = FALSE,orientation = NA,show.legend = NA,inherit.aes = TRUE)
例子:
# import libraries
library("ggplot2")
# import data
df <- ToothGrowth
# Transform dose and len column into factor
dfdose <- as.factor(dfdose)
dflen <- as.factor(dflen)
p<-ggplot(df,aes(len,dose))
p +
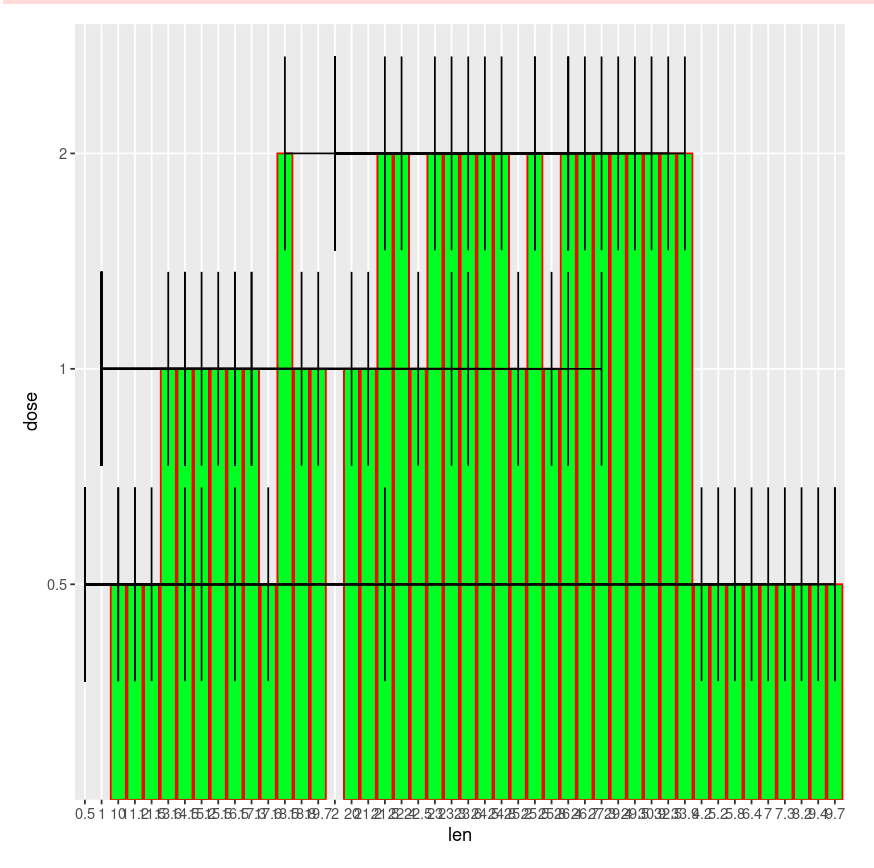
geom_line()+
stat_summary(func.y= mean, geom= "bar" ,
fill="green",color="red") +
geom_errorbarh(aes(xmin =dose , xmax = len)
)
输出:
让我们在一个变量系统上实现这些。
在一个变量中加载你的数据。检查数据是否正确,如果正确则保持不变,否则根据你的需要改变。要绘制电影(x轴)的平均唤醒分数(y轴),首先要创建一个绘图对象。
语法:
factor(x = character(), levels, labels = levels,exclude = NA, ordered = is.ordered(x), nmax = NA)
如果需要改变,通过将它们设置为实际的因子来完成数据转换。然后绘制数据。
例1 :
library("ggplot2")
rawdata <- read.csv("example1.csv", header = TRUE)
rawdatagender = factor(rawdatagender,levels = c(1,2),
labels = c("Men","Women"))
rawdatafilm = factor(rawdatafilm, levels = c(1,2), l
abels = c("Bridget Jones","Memento"))
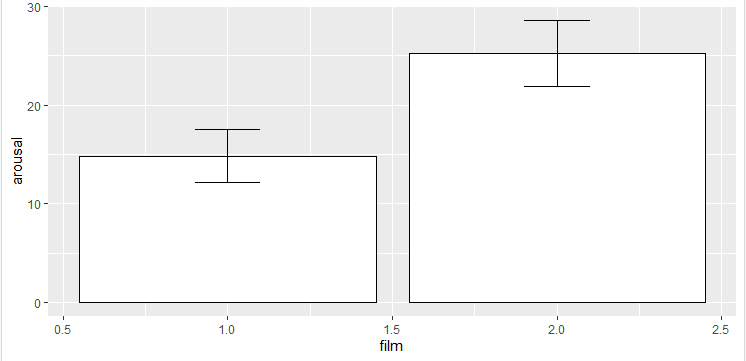
rawdata_bar = ggplot(rawdata,aes(film,arousal))
rawdata_bar + stat_summary(func.y= mean, geom= "bar" ,
fill="white",color="black") +
stat_summary(fun.data = mean_cl_normal ,
geom = "errorbar",width = 0.2)
rawdatagender = factor(rawdatagender,levels = c(1,2),
labels = c("Men","Women"))
rawdatafilm = factor(rawdatafilm, levels = c(1,2),
labels = c("Bridget Jones","Memento"))
rawdata_bar + stat_summary(func.y= mean, geom= "bar" ,
fill="white",color="black")
rawdata_bar + stat_summary(func.y= mean, geom= "bar" ,
fill="white",color="black") +
stat_summary(fun.data = mean_cl_normal ,
geom = "errorbar",width = 0.2)
输出
例2: 对两个自变量也可以这样做。
library("ggplot2")
rawdata <- read.csv("example1.csv", header = TRUE)
rawdatagender = factor(rawdatagender,levels = c(1,2),
labels = c("Men","Women"))
rawdatafilm = factor(rawdatafilm, levels = c(1,2),
labels = c("Bridget Jones","Memento"))
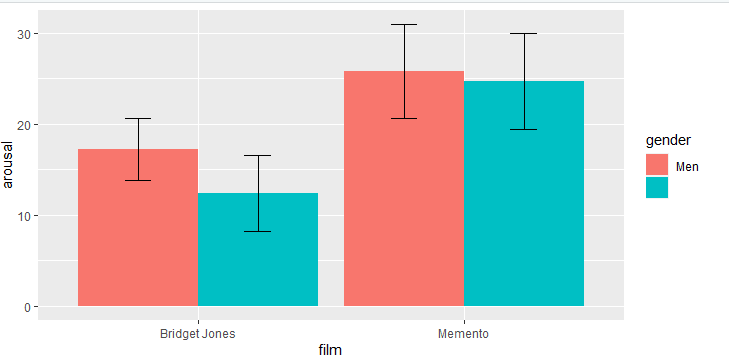
rawdata2 = ggplot(rawdata,aes(film, arousal, fill = gender))
rawdata2 +
stat_summary(fun.y = mean, geom = "bar",position = "dodge")+
stat_summary(fun.data = mean_cl_normal, geom = "errorbar",
position = position_dodge(width = 0.90),width=.2)
输出
 极客教程
极客教程